DNA sequencing
AL DEZE INFORMATIE KOMT VAN DE site van :The University of Michigan's DNA Sequencing Core, voor de duidelijkheid is het hier vertaald
Het sequencen verloopt als in de PCR reactie is beschreven
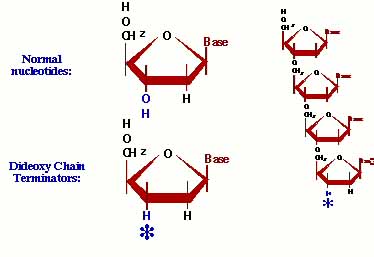
Maar nu laten we de reacties verlopen in de aanwezigheid van een (klein percentage) dideoxyribonucleotides. Deze lijken op een normaal nucleotide, maar hebben geen 3' hydroxyl group , waardoor ze bij een DNA – replicatie wel gekoppeld worden aan het eind van een (groeiende) DNA-keten, maar er tegelijk de oorzaak van zijn dat de keten niet verder verlengd kan worden. Ze vormen zo het uiteinde van de keten
|
De clou is nu dat de meeste nucleotides van het normale type zijn en slechts een kleine minderheid dideoxy nucleotides. Bij het repliceren van een DNA keten in de aanwezigheid van dideoxy-T wordt meestal wanneer een T nodig is in de keten een “goede” T gebruikt door het enzym en zal het enzym daarna gewoon verder gaan met het verlengen van de keten door meer nucleotides toe te voegen. Echter in 5% van de gevallen krijgt het enzym te maken met een dideoxy-T, and die streng kan nooit meer verlengd worden. Uiteindelijk valt de onafgemaakte keten van het enzym. Vroeger of later zullen alle kopieën afgesloten worden met een T, maar elke keer dat het enzym een nieuwe keten maakt is het punt waar gestopt wordt willekeurig. Bij de miljoenen starts zullen er ketens zijn die op elke mogelijke T plaats gestopt zijn . Dus alle mogelijke T-eindpunten komen aan de beurt. Alle ketens zijn op precies dezelfde plaats begonnen en allemaal eindigen ze met een T. Er zijn biljoenen ketens en miljoenen per mogelijke T-positie. Om uit te vinden waar alle T liggen in onze nieuwe keten, moeten we alleen weten wat de lengtes zijn van al onze afgebroken ketens.

Hoe komen we de lengte van deze fragmenten te weten?
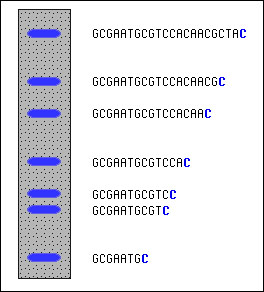
Gel electroforese kan gebruikt worden om fragmenten op grootte te scheiden en te meten. Hierbij zijn de dideoxy nucleotides chemisch zodanig veranderd dat ze fluoresceren onder UV licht. Dideoxy-C, bijvoorbeeld zendt blauw licht uit. Als je nu het reactiemengsel op een electroforesegel brengt zie het volgende beeld: De kleinste fragmenten beneden, de grootste bovenaan. De plaats en afstand geeft de relatieve grootte weer. Beneden is het kleinste fragment dat beëindigd is door ddC; dit is waarschijnlijk de C die het dichtste bij het eind van de the primer ligt (die is weggelaten is op plaatje). Door de gel verder te volgen, zien we dat we twee plaatsen overslaan, dan komen we twee C”s achterelkaar tegen, weer een plaats overslaan en daar is weer een C. enzovoort. Zo kunnen we zien waar alle C’s liggen.
Nu met vier dideoxy nucleotides (A, G, C and T)
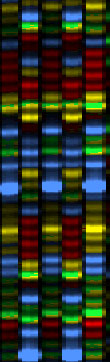
In de praktijk is het niet zo eenvoudig als op het plaatje te zien is. De ruimte tussen de bandjes is niet altijd even gemakkelijk in basenplaatsen uit te drukken. Stel je echter eens voor, dat we de replicatie hebben uitgevoerd met alle dideoxy nucleotides (A, G, C and T) die aanwezig zijn, en elk met een andere fluorescerende kleur. Als je nu kijkt naar de gel hiernaast dan is de volgorde ( sequence) van het DNA tamelijk voor de hand liggend als je de kleurcodes weet : lees de kleuren van beneden naar boven: TGCGTCCA-(etc). (voor de duidelijkheid is geel vervangen door zwart) |
|
Dat is precies wat we doen bij DNA -sequencing, we runnen DNA replicatie reacties in buisje, in de aanwezigheid van kleine hoeveelheden van alle vier dideoxy terminator nucleotides. Een gel scheidt the gevormde fragmenten op grootte and we kunnen de volgorde van beneden naar boven aflezen. Ook hoeven we zelf de kleurtjes niet meer te “lezen”, de computer doet dat voor ons! Hieronder een voorbeeld van wat de computer ons over een monster vertelt: het is de plot (uitdraai, print) van de kleurvolgorde in een monster , gescand van de kleinste tot de grootste fragmenten. De computer zet de kleurcodes om in de letters van de basen. |
|
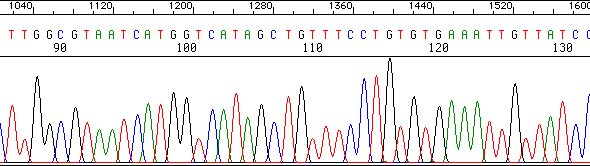
Bron: http://seqcore.brcf.med.umich.edu/doc/educ/dnapr/sequencing.html
Op de site van :The University of Michigan's DNA Sequencing Core